华中农业大学继续教育培训
 咨询电话:
咨询电话:400-0815-589
欢迎来到继续教育培训网院校库!
继续教育培训网首页 咨询电话:
咨询电话:近日,我校动科动医学院赵书红教授团队研发了一款兼具计算高效、内存节省、可视化丰富的全基因组关联分析(Genome-Wide Association Study, GWAS)软件rMVP,其利用分块矩阵计算、并行加速、计算流程优化等策略提升全基因组关联分析的计算效率,采用内存映射技术降低对内存资源的依赖,并提供灵活的基因组大数据处理与可视化模块,为大数据全基因组关联分析研究提供了新的工具。
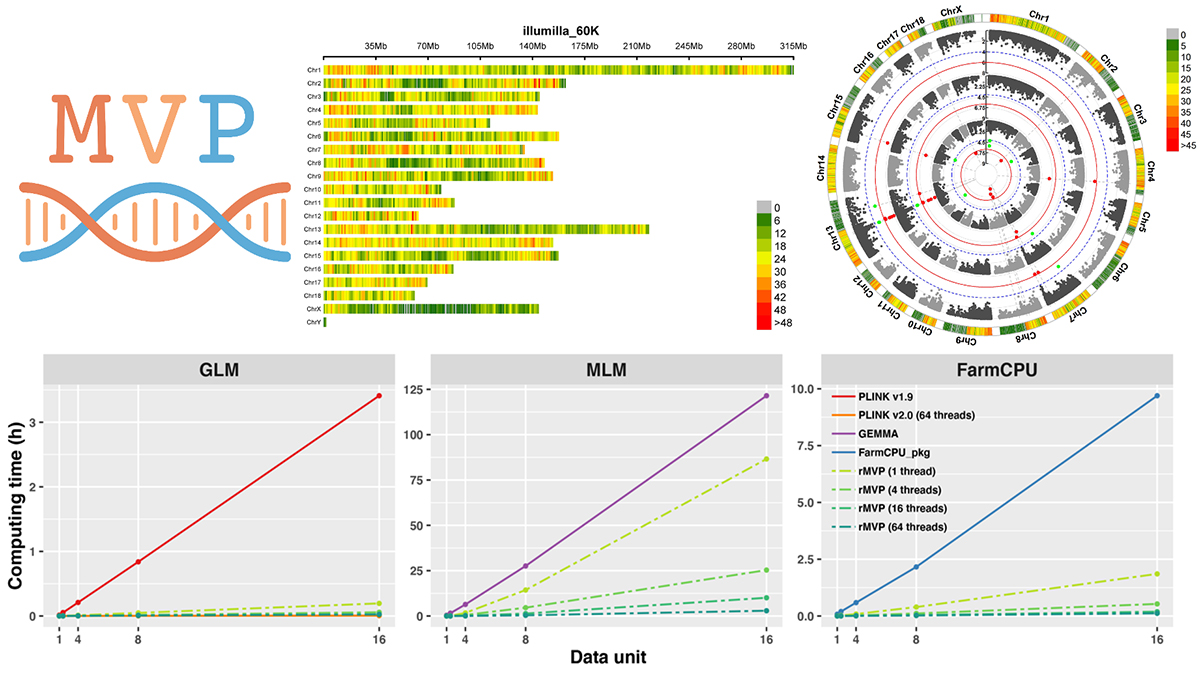
全基因组关联分析是一种通过统计学手段检验表型与覆盖全基因组的高密度标记之间关联程度来挖掘目标性状候选基因的重要方法。近十年来,GWAS被广泛应用于人类、畜牧、水产、植物和微生物等研究领域,鉴定了众多影响人类疾病与农业经济性状的关键基因,成为解析复杂性状遗传机制的一把利器。随着测序成本的降低和表型组技术的发展,用于GWAS的群体数量、标记密度和表型数量迅速增长,尤其对于依赖个体亲缘关系矩阵的混合线性模型,其内存消耗呈现n2级增长(n为个体数量),庞大的数据规模给GWAS带来了新的挑战。因此,如何利用有限的计算资源高效地处理大数据成为目前GWAS领域的研究重点之一。
rMVP软件兼具内存节省(Memory-efficient)、可视化丰富(Visualization-enhanced)、计算高效(Parallel-accelerated)等特点。通过内存映射技术,rMVP软件可直接从磁盘获取数值,有效降低了GWAS分析过程中的内存消耗,同时避免了多进程并行计算时的数据拷贝问题。它采用基于OpenMP技术和Intel MKL技术的“线程级”和“数据级”双并行模式进行计算加速,同时凭借分块矩阵拆分计算、GWAS流程全局优化等策略避免了大矩阵的重复计算,这也使得rMVP比PLINK,GEMMA和FarmCPU_pkg中对应的模型计算速度快约5-20倍。
此外,rMVP软件还开发了一套高质量的可视化工具,可兼容 PLINK,GEMMA,GCTA,TASSEL等软件的分析结果,绘制包括表型分布图、标记密度分布图、群体结构图、曼哈顿图、QQ图在内的多种高分辨率图片。其中,它创新性地采用圆圈式曼哈顿图展示多性状、多模型结果,并首次将标记密度信息引入到曼哈顿图中。
